Como continuação do post Mapas Temáticos usando o R, mostraremos aqui como é possível unir informações pontuais (Point Process) e dados regionais (Lattice Data) além de unir esses dados com o Google maps.
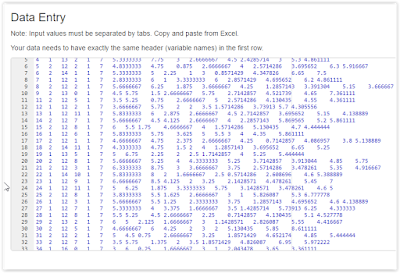
O primeiro passo é a obtenção da malha (shapefile) e dos dados (DadosMapa.csv) para o processo de georeferenciamento.
O primeiro passo é invocar as bibliotecas necessárias bem como definir o Working Directory no qual o arquivo DadosMapa.csv está e também a pasta Shapes
#Limpa o Working Directory
rm(list=ls())
#Define working directory
setwd("C:\\Blog\\ExemploMapas")
#Invoca os pacotes necessários
library(RColorBrewer)
library(maptools)
library(rgdal)
library(rgeos)
library(RgoogleMaps)
library(sp)
library(spdep)
library(ggmap)
library(plyr)
library(Hmisc)
Em seguida, importamos para o R os dados e a malha de interesse:
#Importa os dados
dados<-read.csv("DadosMapa.csv")
#Lê os shapefiles que estão na pasta Shapes dentro do working directory
sfn <- readOGR("Shapes","11MUE250GC_SIR",verbose = FALSE)
Uma vez lidos os dados e malhas, é preciso saber qual a região devemos importar do GoogleMaps, para isso fazemos:
#Bounding box a ser utilizada no ggmap
b <- bbox(sfn)
#Cria uma variável ID para o georeferenciamento
sfn@data$id <- rownames(sfn@data)
#Define a projeção (Shapefile)
sfn <- spTransform(sfn, CRS("+proj=longlat +datum=WGS84"))
#Cria um SpatialPointsDataFrame com as latitudes e longitudes dos dados
spdf <- SpatialPointsDataFrame(coords=dados[,c("Longitude","Latitude")], data=dados)
#Define a projeção para os pontos
proj4string(spdf) <-CRS("+proj=longlat +datum=WGS84 +no_defs +ellps=WGS84 +towgs84=0,0,0")
#Trasnforma o objeto espacial em um objeto que pode ser lido pelo ggplot2
sfn.df <- fortify(sfn, region="CD_GEOCMU")
#Substring a variável ID para ficar com 6 dígitos (ignora o dígito verificador)
sfn.df$V007<-substr(sfn.df$id,1,6)
#Left Join do CSV com o Shapefile
sfn.df<-merge(sfn.df,dados,by="V007",all.x=T)
#Ordena os dados
sfn.df<-sfn.df[order(sfn.df$order), ]
Finalmente, após a união dos dados com o Shapefile podemos construir o mapa. Para isso passamos como argumento do ggmap a bounding box obtida nos passos anteriores e definimos também as cores, escalas, títulos do mapa:
#Aumenta ou diminui a Bounding Box (aumenta em 1%)
bbox <- ggmap::make_bbox(sfn.df$long, sfn.df$lat, f = 0.1)
#Escolhe as cores
myPalette <- colorRampPalette(rev(brewer.pal(11, "Spectral")))
#Obtêm o mapa do Google Maps (Existem outras possibilidades...)
map <- get_map(location=bbox, source='google', maptype = 'terrain', color='bw')
#Constrói o mapa:
map <- ggmap(map, base_layer=ggplot(data=sfn.df, aes(x=long, y=lat)),
extent = "normal", maprange=FALSE)
map <- map + geom_polygon(data=sfn.df,aes(x = long, y = lat, group = group, fill=Valor), alpha = .6)
map <- map + geom_path(aes(x = long, y = lat, group = group),
data = sfn.df, colour = "grey50", alpha = .7, size = .4, linetype=2)
map <- map + coord_equal()
map <- map + scale_fill_gradientn(colours = myPalette(4))
map<-map+ geom_point(aes(x=Longitude, y=Latitude),color="black", size=1,
alpha = 0.70,
data=spdf@data)
map<-map + ggtitle("Postos SINE") + labs(x="Longitude",y="Latitude")
#Plota o mapa
plot(map)
Pode-se ainda definir outras bases para o shapefile, alguns exemplos são apresentados a seguir:
##Pode-se trocar o código: map <- get_map(location=bbox, source='google', maptype = 'terrain', color='bw') ##Do bloco anterior, por algum desses outros: #map <- get_map(location=bbox, source='osm', color='bw')) #map <- get_map(location=bbox, source='stamen', color='watercolor')) #map <- get_map(location=bbox, source='stamen', color='toner')) #map <- get_map(location=bbox, source='stamen', color='terrain')) #map <- get_map(location = bbox, source = 'google', maptype = 'terrain') #map <- get_map(location = bbox, source = 'google', maptype = 'satellite') #map <- get_map(location = bbox, source = 'google', maptype = 'roadmap') #map <- get_map(location = bbox, source = 'google', maptype = 'hybrid')